基因变异分类数据集

文件列表(压缩包大小 3.59M)
免费
概述
转载自https://www.kaggle.com/kevinarvai/clinvar-conflicting
基因变异的分类
ClinVar公共数据集包括关于人类遗传变异的注释。临床实验室采用人工分类的方式对这些变异进行分类,包括良性、可能良性、不确定、可能致病和致病。当临床医生或研究人员研究该变异是否对特定患者的疾病有影响时,分类相冲突的变异可能会对研究造成混淆。
内容
数据集的目的是预测锁骨变异是否有冲突。这是一个二元分类问题,数据集中的每条记录都是一个遗传变量。
当下列三种类型中的任何一种出现时,则认为分类冲突,两次提交同一种分类不冲突。 1 可能良性或良性 2 VUS 3 可能致病或致病
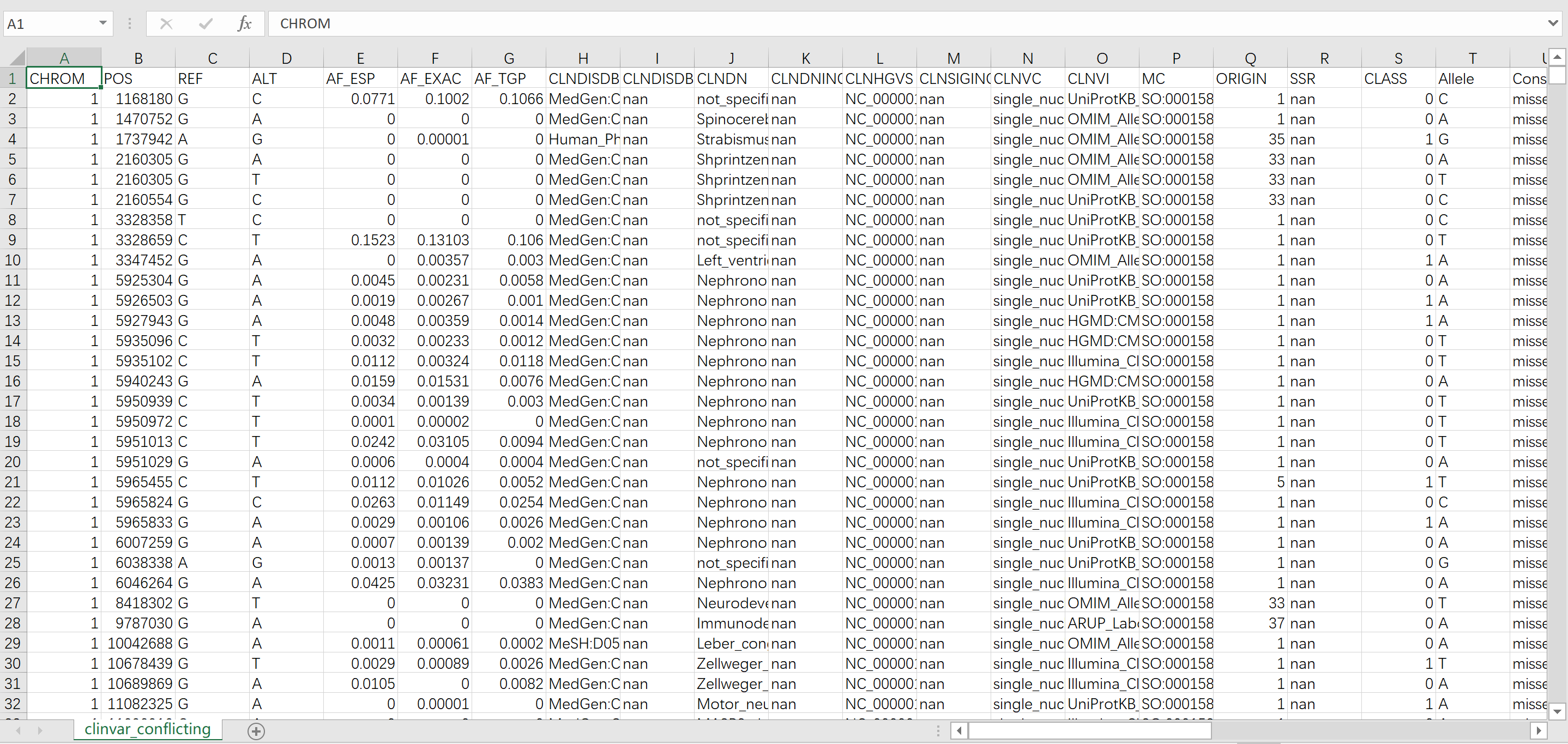
CLASS列代表冲突的分类。它由二进制表示,代表一个变异是否有相互冲突,其中0表示一致的分类,1表示冲突的分类。由于这个问题只与多种分类的变异有关,作者从原来的ClinVar .vcf中删除了所有的变体。 原始变量调用格式(vcf)文件在这里下载: ftp://ftp.ncbi.nlm.nih.gov/pub/clinvar/vcf_GRCh37/clinvar.vcf.gz
致谢
感谢Landrum MJ, Lee JM, Benson M, Brown GR, Chao C, Chitipiralla S, Gu B, Hart J, Hoffman D, Jang W, Karapetyan K, Katz K, Liu C, Maddipatla Z, Malheiro A, McDaniel K, Ovetsky M, Riley G, Zhou G, Holmes JB, Kattman BL, Maglott DR. ClinVar对不同解释和支持的获取和改进。 2018年1月4日 PubMed PMID: 29165669。
激励计划
作者正在探索将机器学习应用于基因组学的可能性。希望这个数据集可以激励其他人考虑其他的功能斌进行相应的工程应用。对尚未分配冲突分类的单次提交进行识别可能会有所提升。
如果遇到文件不能下载或其他产品问题,请添加管理员微信:ligongku001,并备注:产品反馈

评论(0)

 理工酷
理工酷
 资源下载
资源下载